Proteinvielfalt in Bakterien könnte größer sein als gedacht
Ein Team um Matthias Erlacher untersuchte am Institut für Genomik und RNomik an der Med Uni Innsbruck überlappende Leserahmen in bakteriellen mRNAs, welche die Eiweißherstellung in bakteriellen Zellen wesentlich beeinflussen könnten. Auf Basis der neuen Erkenntnisse soll nun u.a. die Frage geklärt werden, ob aufgrund des entdeckten Mechanismus die bakterielle Proteinvielfalt größer ist, als angenommen, oder ob es sich dabei um eine neue Art der Regulation für die Eiweißproduktion handelt.
Proteine, also Eiweiße, werden nach einem Bauplan hergestellt, der auf der DNA gespeichert ist und bei Bedarf in RNA umgeschrieben wird. Diese mRNA wird dann vom Ribosom, der Proteinfabrik der Zellen, abgelesen und in eine Aminosäuresequenz – das heißt, in ein Protein - übersetzt. Damit das Ribosom erkennt, welcher Bereich der mRNA übersetzt werden soll, gibt es so genannte Startcodons und Stopcodons. Diese müssen schnell und exakt erkannt werden, um den Bedarf an Proteinen in einer Zelle zu jedem Zeitpunkt zu decken. Im Rahmen ihrer Forschungsarbeit sind WissenschafterInnen um Matthias Erlacher vom Institut für Genomik und RNomik (Direktor: Alexander Hüttenhofer) auf überlappende Startcodon-Sequenzen in E. coli Bakterien gestoßen, welche das Ribosom vor die Herausforderung stellen, das richtige Startcodon zu erkennen. Je nachdem, bei welchem Startpunkt das Ribosom ansetzt, verschiebt sich der Leserahmen und völlig unterschiedliche Proteine können entstehen. Das bedeutet nun, dass in einer mRNA-Sequenz zwei Eiweiße codiert sein können und die Proteinvielfalt in der (Bakterien-)Zelle möglicherweise größer ist, als bisher angenommen. Die Erkenntnisse wurden kürzlich im Fachjournal Nucleic Acids Research veröffentlicht.
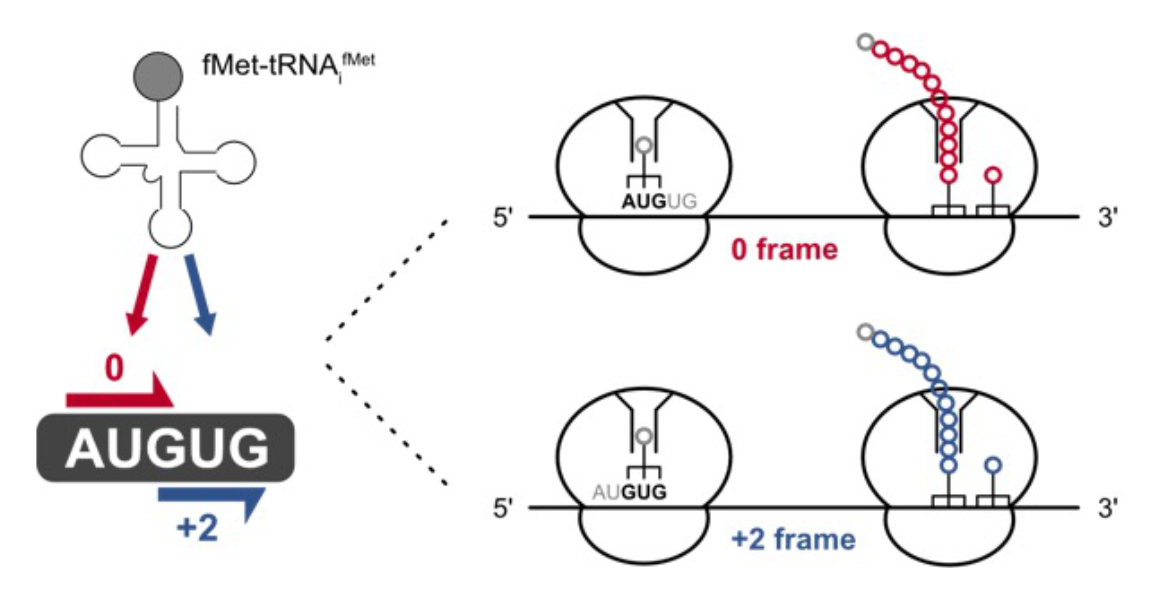 BU: Die Nukleotidsequenz AUGUG in bakteriellen mRNAs kodiert für zwei überlappende Startcodons, die zu unterschiedlichen Proteinprodukten führen können.
BU: Die Nukleotidsequenz AUGUG in bakteriellen mRNAs kodiert für zwei überlappende Startcodons, die zu unterschiedlichen Proteinprodukten führen können.
Das mit Abstand am häufigsten vorkommende Startcodon in allen Organismen ist AUG, eine Abfolge der Nukleotide Adenosin (A), Uracil (U) und Guanosin (G). In bakteriellen mRNAs können auch die Codons GUG und UUG als Startcodon fungieren. Im Zuge ihrer Arbeit in dem vom Forschungsförderungsfond FWF finanzierten Sonderforschungsbereich RNA Deco wurde das Innsbrucker Team auf die Sequenzfolge AUGUG in E.coli mRNAs aufmerksam, die zwei überlappende Startcodons, AUG und GUG, aufweist. Trotz der unmittelbaren Nachbarschaft der zwei Startcodons müssen diese vom Ribosom eindeutig erkannt werden, um das gewünschte Protein herzustellen. „Wir wollten verstehen wie das Ribosom in diesen Fällen die Entscheidung trifft und welche Faktoren diesen Prozess beeinflussen. Dabei stellten wir fest, dass unter bestimmten Vorrausetzungen tatsächlich beide Startcodons erkannt werden. Dies würde letztlich bedeuten, dass ein zweiter Leserahmen in der betreffenden mRNA Sequenz verborgen ist. Folglich ist das bakterielle Proteom, also die Gesamtheit der Proteine, unter Umständen größer als gedacht“, erklärt Erlacher. Maximilian Kohl, ein Student der Molekularen Medizin in Innsbruck, widmete sich in seiner Masterarbeit den daraus entstandenen Fragestellungen und ist Erstautor der nun veröffentlichten Studie.
Zwei wichtige Faktoren bei der Proteinherstellung entdeckt
Den Innsbrucker ForscherInnen ist es gelungen, zwei wesentliche Komponenten zu identifizieren, die die Wahl des Startcodons beeinflussen: die Shine Dalgarno Sequenz (SD) und ein einzelnes Adenin unmittelbar nach dem Startcodon.
Die SD ist ein Sequenzelement, das sehr häufig in bakteriellen mRNAs vor dem Startcodon positioniert ist. Sie ist wichtig für die Rekrutierung und Positionierung der Ribosomen an der mRNA. Die Distanz von SD zum Startcodon ist dabei entscheidend, ob bei AUGUG die Sequenzen AUG oder GUG als Start erkannt werden. Es gibt allerdings auch Gene bei denen gerade aufgrund der Position von SD beide Codons als Start für die Übersetzung verwendet werden.
Die zweite wesentliche Komponente ist ein Adenin, das unmittelbar an das Startcodon anschließt. Dieses Adenin ist in der Hälfte aller bakteriellen Gene zu finden. Bereits bekannt war, dass damit die Effizienz der Eiweißherstellung erhöht wird. Über den dahinterliegenden Mechanismus konnte allerdings nur spekuliert werden. Auch bei den AUGUG Sequenzen konnte man dieses A4 (das A als 4. Nukleotid in der Sequenz, Anm.) identifizieren, woraus sich dann AUGUGA ergibt. „Dadurch entsteht direkt nach dem AUG Startcodon ein UGA Stopcodon. Wenn das Ribosom mit der Übersetzung beim ,falschen‘ AUG startet, dann ist bei UGA gleich wieder Schluss und das Ribosom bekommt die Gelegenheit zu einem Neustart beim ,richtigen‘ GUG Startcodon. Das A ist offensichtlich wichtig, um Fehler am Beginn der Proteinherstellung zu korrigieren. Darüber wurde schon früher, vor allem im Hinblick auf die Evolution des Startcodons, spekuliert. Es war aber noch keine geeignete Methode vorhanden, um das experimentell zu untersuchen. Die überlappenden Startsequenzen gaben uns die Möglichkeit dazu“, sagt Erlacher.
Die Erkenntnisse werfen nun eine Reihe weiterer Fragen auf, welchen die ForscherInnen in künftigen Projekten nachgehen wollen. So ist noch offen, wann und wie weit diese überlappenden Startsequenzen relevant sind. Es gilt herauszufinden, ob die alternativen Proteine tatsächlich eine Funktion erfüllen oder sofort wieder abgebaut werden. Es besteht auch die Möglichkeit, dass die überlappenden Startsequenzen eine regulatorische Funktion haben, die den Start der Übersetzung modulieren. Wichtig ist auch, die Frage zu klären, ob es das Phänomen der überlappenden Startcodons ausschließlich bei E. coli Bakterien gibt, oder es auch in anderen Bakterien und Organismen auftritt. „Spannend ist aber, dass in der mRNA eventuell mehr Information steckt, als wir bisher gedacht haben“, sagt Erlacher.
(Innsbruck, 1. Februar 2023, Text: T. Mair, Bilder: Institut für RNomik und Genomik, Nucleic Acids Research)
Zu den Personen:
Matthias Erlacher studierte Chemie an der Universität Innsbruck und promovierte 2008 an der Medizinischen Universität Innsbruck. Seit 2012 forscht er mit seinem Team am Institut für Genomik und RNomik zu den unterschiedlichsten Aspekten der Proteinbiosynthese. 2020 folgte die Habilitation in Molekularbiologie. Derzeit sind vor allem modifizierte RNAs und deren Rolle in der Zelle im Fokus seiner Forschungsarbeit.
Maximilian Kohl, der Erstautor der Studie, studierte Molekulare Medizin an der Medizinischen Universität Innsbruck und erforscht nun in seiner PhD Arbeit die Translation in Staphylococcus aureus an der Universität Straßburg.
Forschungsarbeit:
Maximilian P Kohl, Maria Kompatscher, Nina Clementi, Lena Holl, Matthias D Erlacher, Initiation at AUGUG and GUGUG sequences can lead to translation of overlapping reading frames in E. coli, Nucleic Acids Research, Volume 51, Issue 1, 11 January 2023, Pages 271–289, https://doi.org/10.1093/nar/gkac1175




