Wie SARS-CoV-2 menschliche Zellen austrickst und was wir daraus lernen können
Auch wenn die Corona-Pandemie nach Einschätzung der WHO offiziell beendet ist, bleibt die Forschung am SARS-CoV-2 Virus weiterhin ein wichtiges Thema. Ein interuniversitäres ForscherInnenteam von fünf Instituten der Medizinischen Universität Innsbruck, unter Federführung von Alexander Hüttenhofer, hat nun herausgefunden, wie das Virus gezielt genetische Abläufe in menschlichen Lungenzellen beeinflusst.
Das Coronavirus nimmt nicht nur über bekannte Protein-codierende Gene Einfluss auf die genetischen Vorgänge in Lungenzellen, sondern auch über sogenannte nicht-codierende RNAs (ncRNAs). Diese kleinen RNA-Moleküle regulieren protein-codierende Gene, indem sie als eine Art „Genschalter“ fungieren und dadurch das Verhalten einer Zelle grundlegend verändern. Die Ergebnisse erschienen nun im renommierten Fachjournal Molecular Therapy – Nucleic Acids.
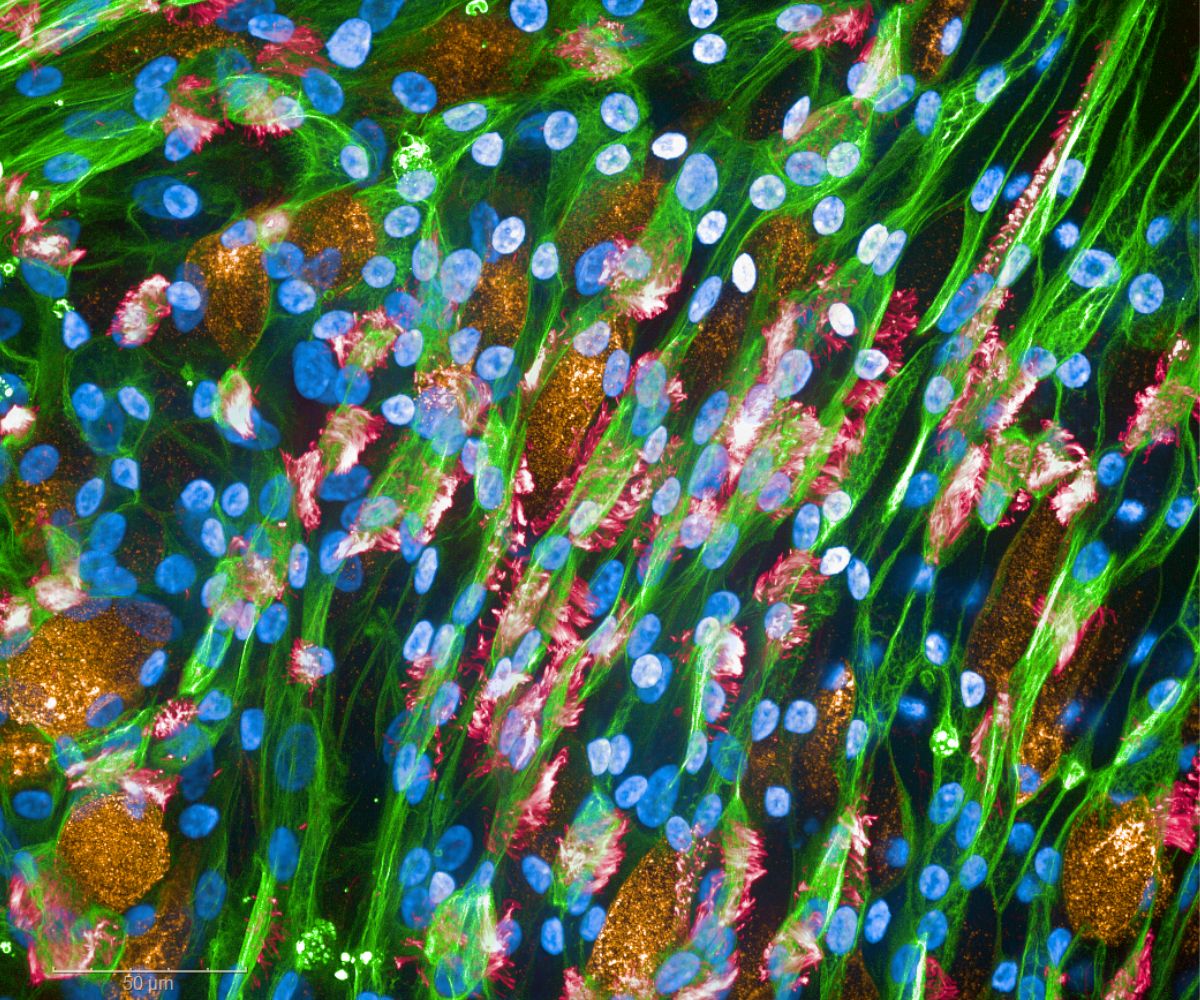
Lungenepithelzellen infiziert mit SARS-Cov2 (Bild: Doris Wilflingseder)
Das interdisziplinäre Forschungsteam verwendete dabei ein besonders realistisches, standardisiertes und optimiertes Lungenzellmodell, das von der Expertin für Zellkulturen, Doris Wilflingseder, noch in Innsbruck entwickelt wurde. Wilflingseder ist mittlerweile Professorin für Infektiologie an der Veterinärmedizinischen Universität Wien und am universitätsübergreifenden Ignaz Semmelweis Institut. Mit dem Modell legte sie einen Grundstein für diese Forschungsarbeit. Die ForscherInnen untersuchten mit diesem Zellmodell, wie sich die Virusvarianten Delta und Omicron auf die Genaktivität von Lungenzellen auswirken. Während sich sowohl eine Stunde als auch 24 Stunden nach Infektion kaum Unterschiede zu nicht infizierten Kontrollzellen zeigten, war drei Tage nach der Infektion das genetische Gleichgewicht der Zellen deutlich gestört – sowohl bei den Protein-codierenden Genen als auch bei den ncRNAs.
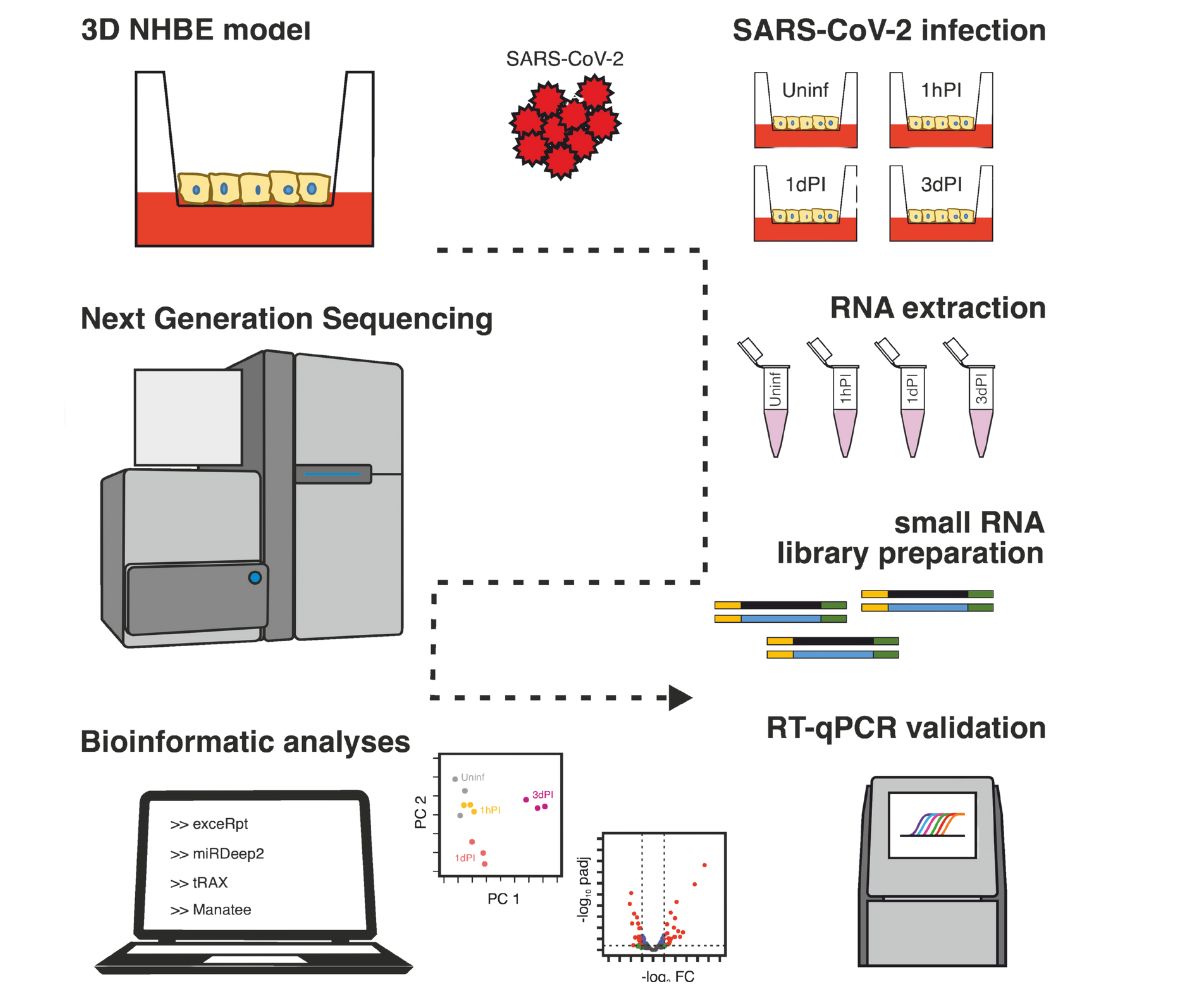
Identifizierung von regulatorischen ncRNAs bei SARS-Cov2 Infektionen (Graphical Abstract: Molecular Therapy – Nucleic Acids/A. Hüttenhofer)
Fünf zentrale Signalwege und deren Auswirkungen wurden dabei in der Studie identifiziert:
1. Angeborene Immunantwort und Typ-I-Interferon-Antwort
2. Virusanheftung, -zusammenbau und –replikation (Vervielfältigung)
3. Entzündungshemmende Signalwege
4. Genregulation und Proteinsynthese (Proteinherstellung)
5. Zellproliferation (Zellvermehrung), Anti-Apoptose (Anti-programmierter Zelltod) und Onkogenese (krankhaftes Zellwachstum)
Durch die Beeinflussung dieser Signalwege war das SARS-CoV-2 Virus in der Lage, infizierte Zellen „am Leben zu halten“, das heißt den programmierten Zelltod (die sogenannte Apoptose) zu verhindern, um sich dadurch besser vermehren zu können. Dabei nutzte es nicht nur Protein-codierende Gene, sondern insbesondere auch bestimmte ncRNA Gene.
Interessant war dabei, dass die ForscherInnen sehr unterschiedliche Reaktionen von Zellen nach Exposition mit Delta oder Omicron bei Protein-codierenden Genen, wie CXCL10, IFIT2 und ZC3HAVI feststellten, jedoch eher moderate Unterschiede zwischen beiden Virusvarianten bezüglich der ncRNA Expression zu beobachten waren. Somit greifen beide Virusvarianten erfolgreich auf ähnliche ncRNA Genen zurück, um ihre Vermehrung zu gewährleisten. Eine Ausnahme davon war, dass das Gen für eine sehr kleine RNA, die zur Klasse der sogenannten microRNAs gehört, die miR-155-5p, bei der Omicron-Variante wesentlich geringer exprimiert war als bei der Delta-Variante. Die miRNA-155 ist dabei als ein wichtiger Genschalter bekannt, der die Immunantwort von B- und T-Zellen steuert. Zudem wurde auch beobachtet, dass in Omicron infizierten Zellen eine fragmentierte Transfer-RNA, die sogenannte tRFGlu(TTC), erhöht vorgefunden wurde. Die unterschiedliche Expression dieser beiden zentralen ncRNA Genschalter könnte möglicherweise also damit zusammenhängen, dass viele Omicron Infektionen milder verlaufen als Infektionen mit der Delta Variante. Somit könnten diese beiden ncRNA Gene in Zukunft sogar als Biomarker dienen, um schwere Krankheitsverläufe frühzeitig zu erkennen.
Wichtige Erkenntnisse für Diagnose und Therapie
Die Studie liefert damit wichtige Erkenntnisse für neue Diagnose- und Therapieansätze. Dabei war besonders spannend zu beobachten, dass die Virusinfektion nicht nur bekannte Gene verändert, sondern auch die Aktivität winziger RNA-Moleküle. Genau diese ncRNAs könnten damit künftig gezielt genutzt werden, um Virus-induzierte ncRNAs – etwa in Exosomen – zur Diagnostik entzündlicher Erkrankungen und für eine Risikostratifizierung bei COVID-19 PatientInnen einzusetzen (i.e. „Liquid Biopsy“).
Aber auch der integrierte methodische Ansatz hebt sich von anderen bisher publizierten SARS-CoV-2 Arbeiten ab, da in dieser Studie durch die interdisziplinäre Zusammenarbeit nicht nur ncRNAs sequenziert wurden (unter Mithilfe der „MultiOmics Core Facility“ der Medizin Uni Innsbruck, Leiterin Anne Krogsdam), sondern die Aktivität der untersuchten Gene zusätzlich durch Methoden wie RT-qPCR oder Northern blot Analysen verifiziert werden konnten. Somit konnte erstmalig ein detaillierter Einblick in die Regulation von kurzkettigen ncRNAs und Protein-codierenden Genen in Form von fünf Signalwegen erhalten werden, der aufzeigt, wie Viren die Immunantworten von infizierten Zellen manipulieren können. Das Verständnis von Veränderungen der ncRNAs während der Virusinfektion wird daher die Auswahl der richtigen RNA Klassen erleichtern, die als Ansatzpunkte für neuartige Therapien geeignet sind, um damit die virusbedingte Replikation zu überwinden und die angeborene Immunantwort nach dem Virus-Eintritt zu unterstützen.
Diese umfassende Studie konnte dabei nur durch die intensive Zusammenarbeit von fünf verschiedenen Instituten an der Medizinischen Universität Innsbruck durchgeführt werden. Das Team bestand dabei aus: Glory Ranches, Erstautorin der Studie, die derzeit am Institut für Neuropathologie und Neuromolekularpathologie beschäftigt ist. Ein Großteil des experimentellen Teils der Studie wurde am Institut für Genomik und RNomik, am Biozentrum Innsbruck unter der Mitwirkung von Melanie Ploner in der Arbeitsgruppe von Alexander Hüttenhofer durchgeführt. Hubert Hackl vom Institut für Bioinformatik am Innsbrucker Biozentrum war für bioinformatische Analysen verantwortlich. Weiters wirkten Viktoria Zaderer und Wilfried Posch vom Institut für Hygiene und Medizinische Mikrobiologie mit. Die enge Zusammenarbeit folgender Personen war dabei essentiell für den Erfolg des ambitionierten Projektes: Doris Wilflingseder, Kai Kummer, Assistenzprofessor am Institut für Physiologie, war entscheidend für die Analyse und Integration der experimentellen Daten und Alexander Hüttenhofer vom Institut für Genomik und RNomik, Biozentrum Innsbruck, initiierte die vorliegende Studie.
Forschungsarbeit: Glory Ranches, Hubert Hackl, Viktoria Zaderer, Melanie Ploner, Wilfried Posch, Doris Wilflingseder, Kai Kummer, Alexander Hüttenhofer, Differentially expressed ncRNAs as key regulators in infection of human bronchial epithelial cells by the SARS-CoV-2 Delta variant, Molecular Therapy Nucleic Acids, Volume 36, Issue 2, 2025, 102559, ISSN 2162-2531, https://doi.org/10.1016/j.omtn.2025.102559.
(Innsbruck, 29. 7. 2025, Text: S. Prock, Foto: Alexander Hüttenhofer, Doris Wilflingseder)
Weiterführende Links:
Institut für Genomik und RNomik
Institut für Hygiene und Medizinische Mikriobiologie




